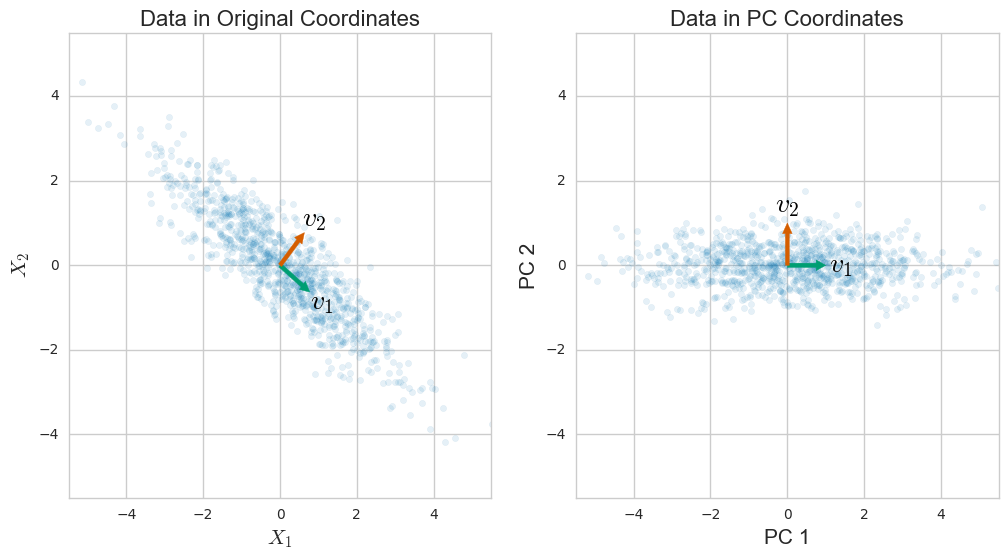
Zacznę od PCA. Załóżmy, że masz n punktów danych składających się z d liczb (lub wymiarów) każdy. Jeśli wyśrodkujesz te dane (odejmij średni punkt danych od każdego wektora danych ), możesz ułożyć dane w stos, aby utworzyć macierzμxi
X=⎛⎝⎜⎜⎜⎜⎜⎜xT1−μTxT2−μT⋮xTn−μT⎞⎠⎟⎟⎟⎟⎟⎟.
Macierz kowariancji
S=1n−1∑i=1n(xi−μ)(xi−μ)T=1n−1XTX
miary, w jakim stopniu różne współrzędne, w których podane są dane, różnią się razem. Nic więc dziwnego, że PCA - który jest zaprojektowany do przechwytywania zmienności twoich danych - może być podany w postaci macierzy kowariancji. W szczególności okazuje się , że rozkład wartości własnejS
S=VΛVT=∑i=1rλivivTi,
gdzie to ty główny składnik lub PC, a to wartość własna i jest również równa wariancji danych wzdłuż komputera. Rozkład ten pochodzi z ogólnego twierdzenia z algebry liniowej, a niektóre prace nie muszą być wykonane, aby motywować relatino do PCA.viiλiiSi
SVD to ogólny sposób na zrozumienie macierzy w kategoriach przestrzeni kolumn i przestrzeni wierszy. (Jest to sposób na przepisanie dowolnej macierzy w kategoriach innych macierzy z intuicyjnym stosunkiem do przestrzeni wierszy i kolumn.) Na przykład dla macierzy możemy znaleźć wskazówki i w domenie i zakresie, abyA=(1021)uivi
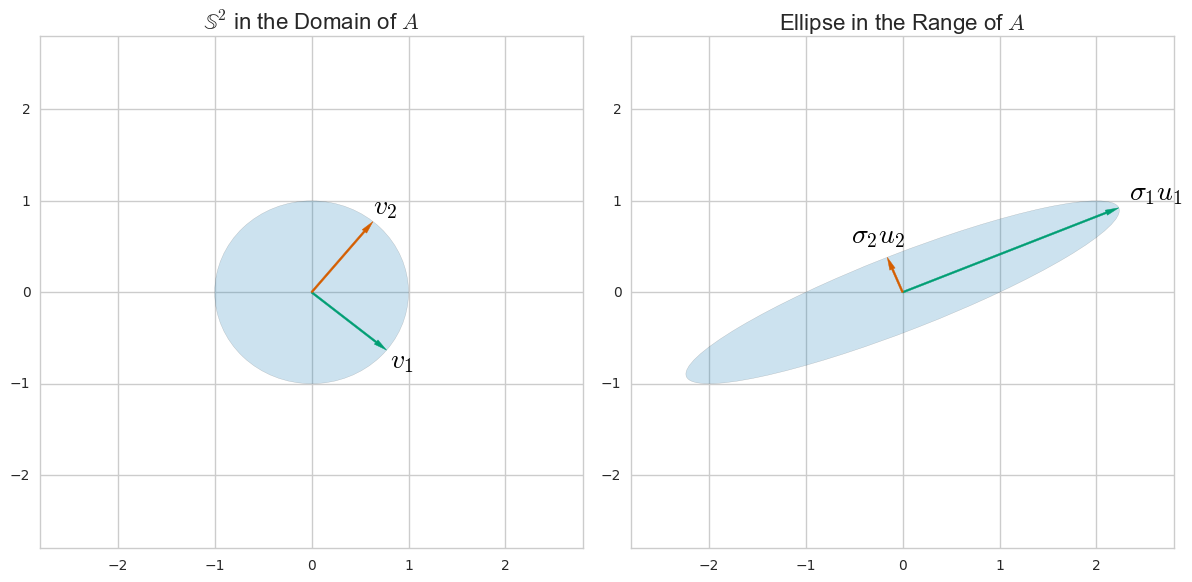
Można je znaleźć, biorąc pod uwagę, w jaki sposób jako transformacja liniowa przekształca sferę jednostkową w swojej dziedzinie w elipsę: główne półosie elipsy wyrównują się z a są ich pierwowzorami.ASuivi
W każdym razie, dla powyższej macierzy danych (tak naprawdę, po prostu ustaw ), SVD pozwala nam pisaćXA=X
X=∑i=1rσiuivTj,
gdzie i są ortonormalnymi zestawami wektorów. Porównanie z rozkładem wartości własnych ujawnia, że „prawe wektory osobliwe” są równe komputerom osobistym, „prawe wektory osobliwe” są{ v i } S v i{ui}{vi}Svi
ui=1(n−1)λi−−−−−−−−√Xvi,
a „wartości pojedyncze” są powiązane z macierzą danych za pośrednictwemσi
σ2i=(n−1)λi.
Jest to ogólnie to, że odpowiednie pojedyncze wektory obejmują przestrzeń kolumny . W tym konkretnym przypadku daje nam skalowane rzutowanie danych na kierunek głównego składnika. Lewe pojedyncze wektory ogólnie obejmują przestrzeń wierszy , co daje nam zestaw wektorów ortonormalnych, które obejmują dane podobnie jak komputery PC. X u i X i v i XuiXuiXiviX
W tym dłuższym artykule omówię więcej szczegółów i korzyści związanych z relacją między PCA i SVD .


